Research Progress on Tobacco Genomic Assisted Breeding
-
摘要:
优质的烟草品种是促进烟草经济发展的基础。2010年以来,烟草基因组学迅速发展,推动了烟草遗传进化、功能遗传学等多个研究领域的进步,为烟草育种提供了新的工具和途径。本文总结了烟草基因组测序组装和功能研究的最新成果,从烟草遗传连锁图谱构建、数量性状基因座(QTL)定位、全基因组关联分析(GWAS)以及QTL定位和GWAS联合分析4个方面阐述了烟草分子标记辅助选择育种(MAS)的发展现状,并综述了全基因组选择(GS)和基因编辑(GE)的技术理论以及它们在烟草育种中的应用进展。针对目前烟草基因组辅助育种(GAB)存在的问题和不足,指出要充分利用烟草基因组信息,加快分子标记的开发,推进基因芯片的应用和加强高通量表型监测平台的建设,进而推进GAB在烟草育种上的应用。
Abstract:Superior variety is basic for tobacco economic promotion. Since 2010, the rapid development of tobacco genomics led to the progress of tobacco genetic evolution, functional genetics, and provided new tools and approaches for tobacco breeding. We summarized the latest achievements in tobacco genome assembly and functional gene, described the current development of tobacco molecular marker-assisted selective breeding (MAS) involving construction of tobacco genetic linkage map, quantitative trait locus (QTL) localization, genome-wide association analysis (GWAS), and the joint analysis of QTL localization and GWAS. We also reviewed the technical theories of genome-wide selection (GS) and gene editing (GE) and their application progress in tobacco breeding. To improve the tobacco genome-assisted breeding (GAB), it is necessary to make full use of the information of tobacco genome, accelerate the development of molecular markers, promote the application of gene chips, and strengthen the construction of high-throughput phenotype monitoring platforms, so as to promote the application of GAB in tobacco breeding.
-
烟草是世界上重要的经济作物之一,病虫害以及各种不利外界环境会影响烟草产量和品质[1-2],且烟草中含有尼古丁等危害人类健康的物质[3]。培育具有高产、高质量、高抗性、低尼古丁含量等特点的烟草新品种是解决以上问题最有效的方法。
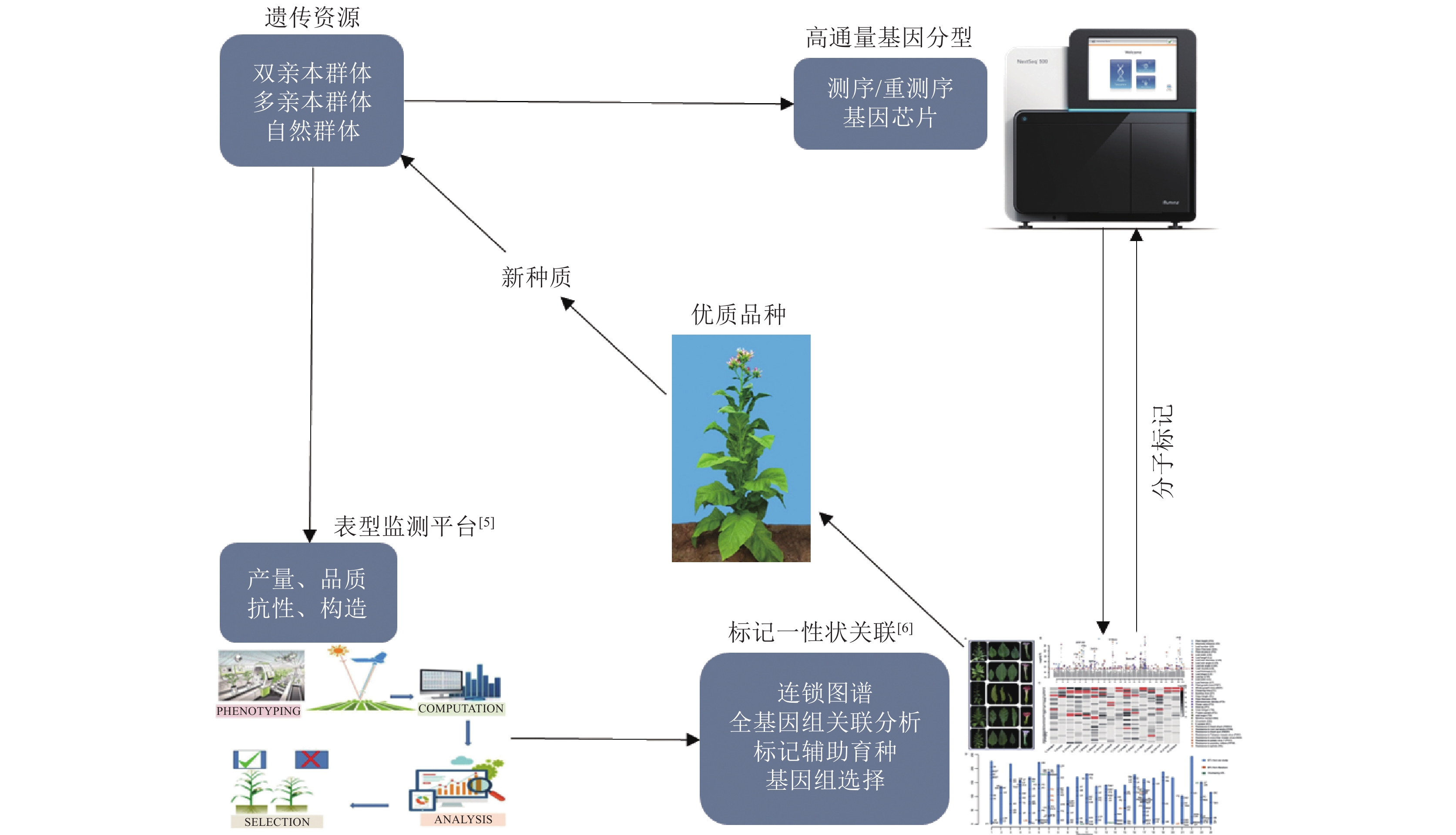
传统的烟草育种主要依赖基于经验的表型选择,虽取得了良好的育种效果,但劳动强度大、耗时长、选择效率低且过度依赖环境。基因组辅助育种(GAB)具有精度高、育种周期短、选择效率高等优点,使育种者能够从大量具有某基因型特征的子代开始,仅使用选定的子集进行更精准的表型评估,找到具有最佳的等位基因组合、最佳的基因网络和特定基因组区域的个体进行繁育,从而实现作物改良(图1)。GAB包括分子标记辅助选择(MAS)、全基因组选择(GS)和基因编辑(GE)等以基因组信息为基础的育种技术。MAS通过标记选择达到对性状定向选择的目的,能够在早期世代中富集有利的等位基因,显著缩短了育种年限,提高了对后代群体选择的准确性,成为了目前主流的育种技术[7-8]。在烟草育种上已经开发了许多MAS策略,包括标记辅助回交(MABC)[9]和标记辅助基因聚集(MAGP)[10]。烟草基因组测序组装的完成以及高通量测序技术的发展,使得高密度的单核苷酸多态性(SNP)标记能够以非常低的成本对烟草进行全基因组分析[11],并且可以精确地将烟草基因型和表型进行关联分析,促使了GS在烟草育种上的应用[12]。GS通过高密度全基因组SNP对个体基因组育种值(GEBV)进行估计,使得对后代群体的选择更加精确。以CRISPR/Cas9为代表的GE技术可以有效针对某一基因对作物进行定向改良,已在烟草品质[13]、产量[14]和抗逆性[15]的定向改良上尝试应用,并取得了一定进展。本文系统总结了烟草基因组研究、分子标记辅助选择(MAS)、全基因组选择(GS)和基因编辑(GE)方面的最新进展,并分析了这些技术在烟草GAB的重要性和应用前景,以期为烟草遗传改良提供理论参考,为加快烟草优良品种的精准选育提供新思路。
![]() 图 1 烟草基因组辅助育种流程图[4]Figure 1. Flowchart for genome-assisted breeding in tobacco
图 1 烟草基因组辅助育种流程图[4]Figure 1. Flowchart for genome-assisted breeding in tobacco1. 烟草基因组研究
2010年以来,高通量测序技术的发展使烟草基因组学发展迅速。目前主要以烟草基因组的序列结构研究为出发点,通过功能缺失型筛选和功能获得型筛选两大策略系统地开展功能基因组学研究,进而帮助育种工作者确定目标基因的功能和作用机制,以便更好地选择和利用这些基因进行育种。
1.1 基因组测序与组装
烟草属于茄科烟草属,已知有66个种,不同种的基因组之间差异较大。世界上最大的栽培种是普通烟草(Nicotiana tabacum,2n=48),它由林烟草(Nicotiana sylvestris,2n=24)和绒毛状烟草(Nicotiana tomentosiformis,2n=24)杂交后染色体自然加倍进化而来[16]。目前已经有多个烟属物种的基因组完成了测序组装。
中国于2011年完成了绒毛状烟草、林烟草和普通烟草全基因组测序组装工作,绒毛状烟草和林烟草的基因组大小为2.4 Gb,contig N50均超过20 kb,scaffold N50均超过350 kb,普通烟草基因组大小为4.4 Gb,contig N50和scaffold N50分别达到为41.6 kb和1.62 Mb[17]。Sierro等[18-19]在2013年至2014年间也陆续公布了绒毛状烟草、林烟草以及普通烟草的多个栽培品种的测序组装结果。Kurotani 等[20]使用HiFi Reads技术对本氏烟(Nicotiana benthamiana)全基因组进行了从头组装,基因组大小为3.1 Gb,contig N50达到141.8 Mb,解码了本氏烟95.6%的基因组信息。野生种渐狭叶烟草(Nicotiana attenuata)和欧布特斯烟草(Nicotiana obtusifolia)的基因组信息报道于2017年[21],渐狭叶烟草基因组大小为2.5 Gb,contig N50和scaffold N50分别为90.4 kb和524.5 kb,欧布特斯烟草基因组大小为1.5 Gb,contig N50和scaffold N50分别为59.5 kb和134.1 kb。其后,黄花烟草(Nicotiana rustica)、波缘烟草(Nicotiana undulata)、圆锥烟草(Nicotiana paniculata)和奈特氏烟草(Nicotiana knightiana)的基因组被成功组装,基因组大小分别为5.0 Gb、2.2 Gb、3.3 Gb和3.1 Gb,scaffold N50分别为84.6 kb、61.9 kb、52.8 kb和82.7 kb[22]。大量烟草基因组序列的发表为烟草基因功能的研究提供了宝贵的基础信息,推动了烟草功能基因组学的发展。
1.2 功能基因
烟草功能基因的表达水平和功能发挥与其生长发育、细胞分化及代谢过程密切相关,深入研究烟草功能基因的表达、调控及互作机制,对于构建烟草生长发育及代谢过程的分子调控网络具有重要意义。NtCXE22在烟草中通过调节独脚金内酯(SLs)的代谢调控腋芽生长,敲除NtCXE22的烟草SLs含量降低、腋芽数量减少且缩短[23]。过表达NtabDOG1L的烟草根系生长能力和抗旱能力得到提升[24]。Mao等[25]通过构建NtCBL4A-1过表达和敲除个体,在不同浓度NaCl处理下观察其农艺性状和生理性状的变化,发现过表达个体对Na+诱导的盐胁迫更加敏感。Zhang等[26]从抗根结线虫(RKN)的烟草品种TI706中克隆了抗性基因NtRk1,发现NtRk1在易感栽培品种“长脖黄”中的过表达增强了其对南方根结线虫(M. incognita)的抗性,在抗性栽培品种K326中对NtRk1进行RNA干扰则导致其对M. incognita具有易感性。NtMYC2a在烟草烟碱生物合成和运输的调控中起“开关”的作用[27]。A622编码的酶与烟草的生物合成有关,Burner等[28]基于CRISPR/Cas9的编辑技术获得A622敲除品系,发现敲除品系在田间和温室试验中烟碱积累均明显减少,但A622的敲除也使得烟草的生长发育受到严重影响,开花延迟,烟叶产量比对照株低60.6%。以上研究从基因层面上解释了烟草在生长发育、抗逆性和生物合成方面的调节机理。随着烟草基因家族全基因组鉴定和功能分析的研究发展,大量的基因家族得到鉴定和分析,控制不同性状的基因家族信息不断得到完善。
2. 分子标记辅助选择育种(MAS)
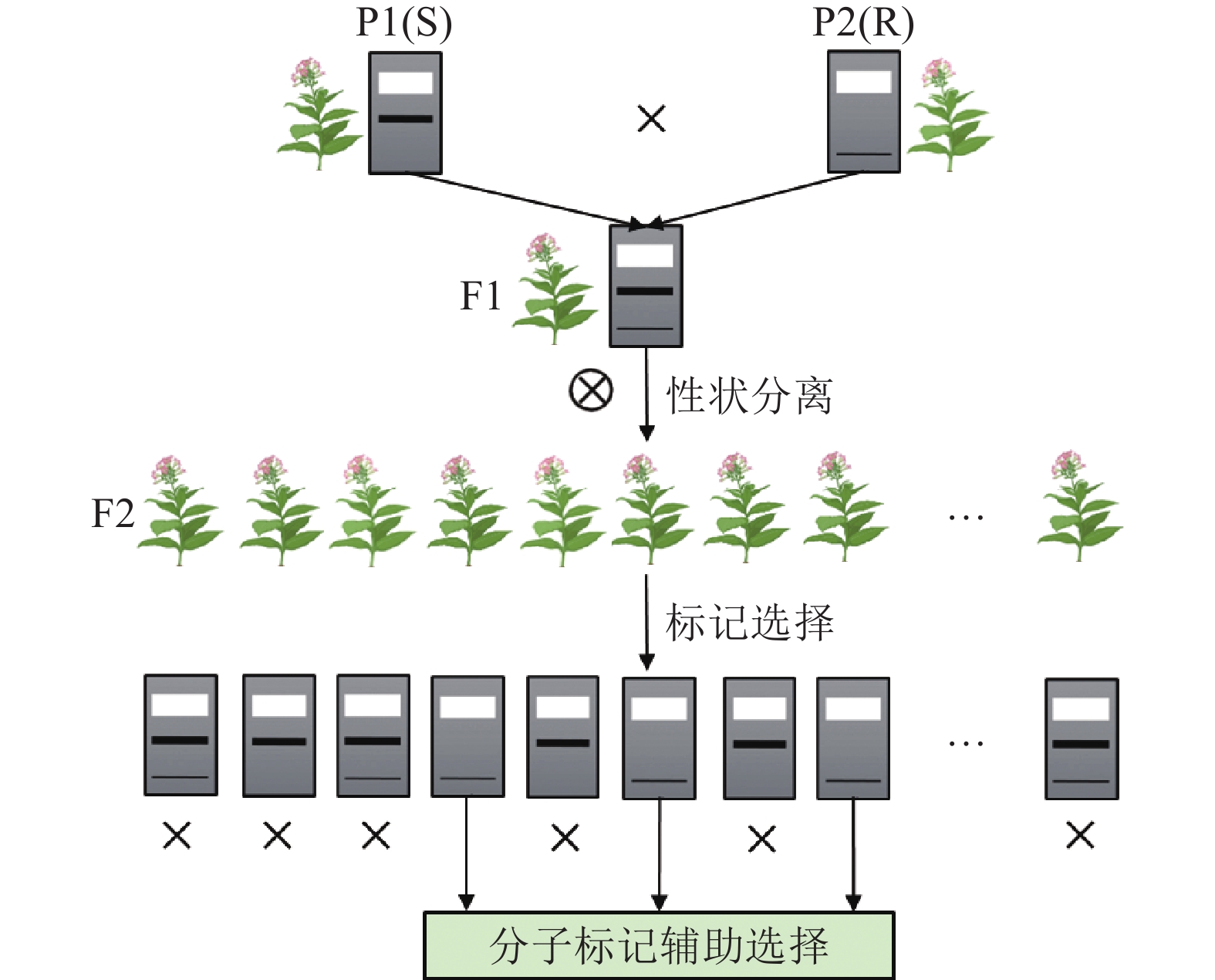
分子标记辅助选择育种(MAS)是一种基于分子标记与目标性状之间的关联性,选择具有所需性状的个体进行育种应用的方法[29](图2)。随着各种分子标记和遗传图谱的出现,MAS不仅在显著的质量性状上发挥作用,在由微效多基因控制的数量性状上也变得可行[30]。数量基因座(QTL)定位和全基因组关联分析(GWAS)是目前MAS标记定位的主要技术手段。
2.1 遗传连锁图谱构建
遗传连锁图谱是分子标记应用于作物遗传育种的基础。Lin等[31]利用野生烟草构建了第一张烟草分子标记遗传连锁图谱,该图谱包括69个RFLP标记和102个RAPD标记。Nishi等[32]利用白肋烟构建了包括95个标记的连锁图谱,这是第一张普通烟草分子标记遗传连锁图谱。为了进一步增加图谱密度,Xiao等[33]基于第二代RAD测序技术的两种不同的SNP筛选方法构建了两个烟草遗传连锁图谱,有2162和4318个SNP定位到24个连锁群,长度分别为2000.9和1944.74 cM。Tong等[34]通过全基因组测序从271个重组自交系(RIL)群体、F1及其亲本中获得3 961 202个高质量SNP,并以K326为参考,对45 081 SNP进行了表征,构建了长度为3 486.78 cM的遗传图谱。烟草遗传图谱的不断完善极大地促进了烟草MAS应用。
2.2 数量基因座(QTL)定位
QTL定位通过将遗传群体中目标性状与遗传标记相结合,根据标记与QTL的连锁关系,确定控制数量性状的遗传区域、大小以及作用强度等信息,主要适用于受控制较强、遗传效应较高的复杂性状[35]。
2.2.1 QTL检测模型
为了鉴定控制烟草复杂性状的遗传变异,已经开发了多种QTL检测模型。Wei等[36]和Li等[37]分别提出了单位点线性混合模型(UPLMM)和多位点线性混合模型(MLLMM)来鉴定MAGIC、NAM和ROAM等多亲群体中复杂性状的QTL;Zhang等[38]发展了完备区间作图法(ICIM),将该模型应用到了对NAM和MAGIC群体的QTL鉴定。上述模型在特定QTL的鉴定中取得了较好的效果。赵会纳等[39]利用ICIM方法鉴定到叶宽相关主效QTL(qMLW20-1),可解释36.8%的表型变异。刘颖超等[40]基于线性混合模型(LMM)进行QTL定位分析,发现了控制总植物碱qTPA14、烟碱qNIC14、假木贼碱qANAB14和新烟草碱qANAT14的4个主效QTL。
2.2.2 烟草QTL定位应用
烟草中已经成功鉴定并克隆了多个与生长发育、胁迫、生物合成相关的QTL。Agacka-Mołdoch等[41]在24个连锁群中发现有11个连锁群包含发芽相关性状的QTL,证明烟草种子萌发相关的性状非常复杂,且受多基因控制。姜自鹏等[42]定位到4个与烟草株高和叶数相关的主效QTL,每个QTL均可以解释相应性状10%~20%的表型变异。蒋勋等[43]基于简化基因组测序发现6个叶面褶皱度QTL、3个支脉粗细QTL、1个主支脉夹角QTL分别在多个环境中被重复检测到。Tong等[44]定位了与自然株高、自然叶数、绝对株高等6个生长发育相关的7个QTL,分别是qLW6-2、qnPH6-2、qnPH6-5、qnLN6-1、qnLN6-3、qSG6-1和qIL6-3。Yuan等[45]鉴定出了与低烟碱特性相关的4个QTL。王思齐等[46]研究中检测到了28个青枯病抗病QTL,有7个QTL在不同调查期中被重复定位,并发现在不同调查期检测到的QTL数目与表达效应存在较大差异,表明在烟草青枯病发病的不同阶段发挥作用的抗性基因可能不同,且其表达具有一定的时序性。Ma等[47]鉴定并克隆了phn7.1,并证明了它与疫霉菌抗性相关。大量QTL的成功鉴定,极大促进了烟草复杂性状的改良进程。
2.3 全基因组关联分析(GWAS)
GWAS是通过对大规模的群体DNA样本进行全基因组高密度遗传标记(如SNP)分型,从而寻找与复杂性状相关的遗传信息的技术方法,主要适用于影响较小、遗传效应较弱、且具有多基因遗传效应的复杂性状。
2.3.1 GWAS分析模型
与QTL作图一样,GWAS也使用遗传标记和目标性状之间的统计关联作图(AM)。多种统计模型[一般线性模型(GLM)、混合线性模型(MLM)、压缩混合线性模型(CMLM)、优化压缩混合线性模型(ECMLM)、循环概率统一的固定和随机模型(FarmCPU)等]被应用于AM,以找到重要的遗传标记[48-49],其中FarmCPU在关联准确性和控制假阳性和假阴性方面都优于其他模型[50]。
2.3.2 烟草GWAS应用
GWAS能在烟草育种中快速定位控制烟草性状的关键基因,进而有效地开展烟草遗传改良工作,提升烟草在产量、品质、抗性等方面的性状表现。孙滢等[51]对烟草开花期典型性状进行GWAS分析,检测到了13个与烟草开花期性状显著关联的SNP,初步确定了控制开花时间的关键候选基因Ntab0279610。Kram等[52-53]利用126 602个SNP,分别对产量性状和株高进行了GWAS分析,检测到与7个产量性状相关的465个数量性状核苷酸(QTN),能够解释0.46%~30.99%的表型变异;并在后续的研究中检测到181个和29个株高相关QTNs,能够解释0.69%~25.37%的表型变异。为了通过育种提高烟叶质量,Tong等[54]检测了与总糖(TS)、还原糖(RS)、总氮(TN)、烟碱(NIC)和总钾(TP)5个化学性状相关的标记,在TS、RS、TN、NIC和TP中分别检测到2、2、4、6和1个显著相关的SNP。Xu等[55]通过GWAS分析,获得了1个与叶片淀粉含量显著相关的SNP位点AX-106011713,并发掘到一个与烟草叶片淀粉含量变异显著相关的候选区域,得到了2个候选基因。青枯病被喻为烟草的“癌症”[56],一直备受关注,Lai等[57]通过GWAS确定了142个与青枯病相关的QTN,能够解释0.49%~22.52%的表型变异。何斌彬等[58]通过GWAS分析,筛选鉴定了8个高抗青枯病的烟草品种,并在烟草基因组内发掘到1个与烟草青枯病抗性变异显著关联的区域,能解释14.29%的表型变异,并预测到4个抗病候选基因。
2.4 QTL定位和GWAS联合分析
GWAS可以快速鉴定关键遗传变异,QTL定位可以确定特定基因影响的数量性状,通过将两种技术整合能够弥补假阳性导致的局限性,有利于检测小效应的QTL,提高检测精度和效率[59]。Zan等[6]利用GWAS和QTL定位对烟草基因组进行联合分析,建立了39个植物形态、发育和抗病性状的参考基因型-表型图谱,发现了一个新的基因Arf9,在敲除后表现出更宽的烟草叶片,并被精确地定位到一个SNP。两种策略的结合促进了烟草在遗传分析和识别候选基因方面的发展,有助于烟草育种工作者更有效地开展烟草遗传改良工作。
3. 全基因组选择(GS)
3.1 GS技术
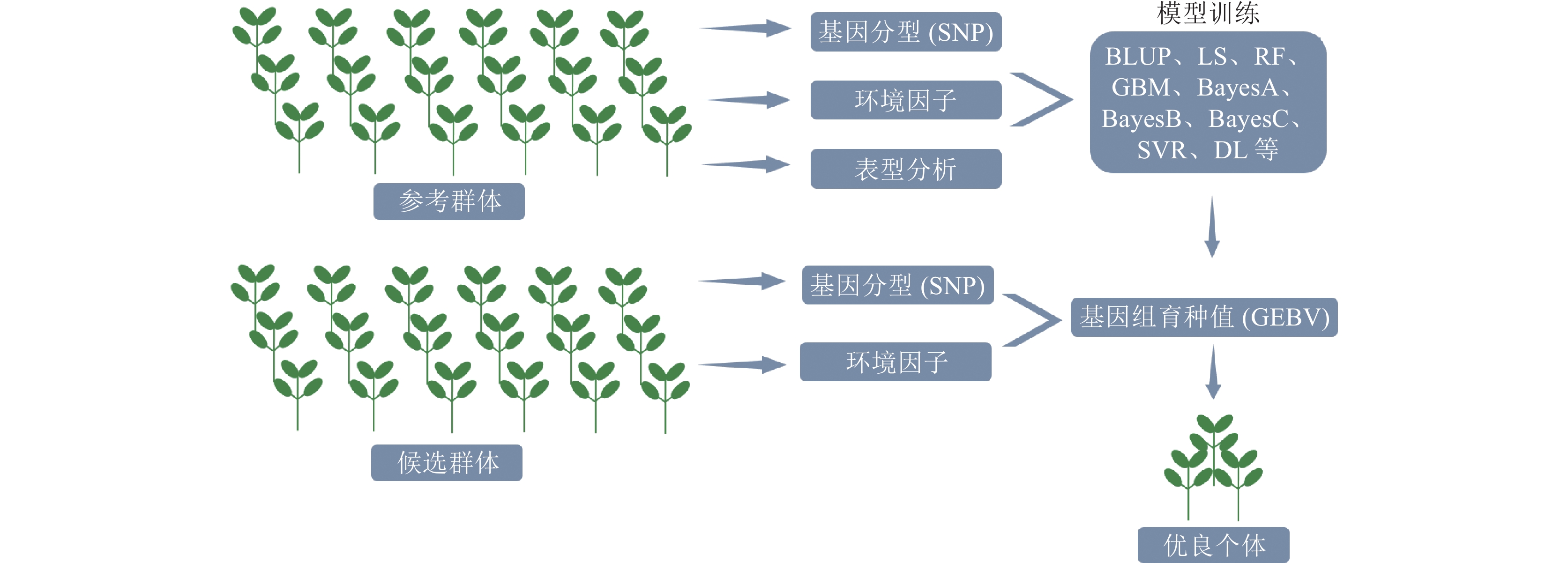
GS是植物育种中一种基于高密度全基因组SNP,结合表型数据对个体基因组育种值(GEBV)进行估计的育种策略[60],包括参考群体和候选群体。对于参考群体,基于高通量测序和基因分型技术获取全基因组SNP,经田间监测平台获取作物的表型信息,结合表型数据估计每个SNP的效应值,构建预测模型;对于候选群体,基于预测模型结合参考群体中获得的SNP效应值累加估算出测试个体的GEBV,选择优良个体[61](图3)。GS的准确性受到GS模型中数据的影响,包括参考群体的大小、参考群体和候选群体之间的关系、标记密度、环境因子等。在参考群体和候选群体中使用亲缘关系较差的个体时,GS的预测准确度下降,参考群体的减少、SNP密度的降低也会导致准确度的降低[62-64]。不同的建模方法对GS预测的准确度和效率影响较大,目前已有多种统计模型应用于GS,包括最佳线性无偏预测(BLUP)、最小二乘法(LS)、贝叶斯法、机器学习(ML)、决策树等。BLUP和贝叶斯法是目前GS的常用方法。Meuwissen等[65]对比了BLUP和LS,BLUP和LS的估计值与真实育种值之间的相关系数分别为0.732和0.318,BLUP明显优于LS。
3.2 烟草GS应用
GS在烟草育种中的应用目前处于试验阶段,Tong等[66]在烟草中进行了GS的试点测试,采用不同的标记数量、群体大小和模型对重组自交系(RIL)群体进行了基因组预测分析,发现群体大小是提高预测准确度的最重要的因素,BayesB模型可能是最适合烟草RIL群体的GS模型。Carvalho等[67]利用13个烟草近交系的72个杂交组合,在两个不同的田间环境中进行了评估,并在不同信息缺失水平下进行预测模型验证,发现基因型与环境的交互作用会影响GS的效率,同时利用基因型和表型信息能提高选择的准确性。随着高通量测序技术的进步、分子标记技术的发展、高通量表型监测平台的建设以及统计模型的改进和完善,GS将成为今后烟草育种的重要工具。
4. 基因组编辑(GE)
4.1 GE技术
GE是一种能够精准对生物体基因组特定基因进行修饰的基因工程技术,其中人工核酸酶[锌指核酸酶(ZFN)、转录激活样效应因子核酸酶(TALEN)和成簇调节间隔短回文重复序列(CRISPR/Cas9)系统]的GE技术可以对目标片段进行靶向编辑,尤以CRISPR/Cas9的应用最广泛,编辑效果最好,已经成功应用于多种作物。CRISPR/Cas系统来源于细菌和古细菌在长期演化过程中形成的一种适应性免疫防御,用来抵御入侵的病毒及外源DNA[68]。CRISPR/Cas9由CRISPR序列、sgRNA和Cas9蛋白三部分组成,sgRNA具有与所选基因组目标序列互补的区域。在sgRNA的指示下,Cas9蛋白定点对靶序列进行切割,产生DNA双链断裂,在对断裂部位修复过程中可能出现点突变、插入、缺失不同类型的变异,使靶序列产生突变,进而达到对基因组进行定点编辑的目的[69]。
4.2 烟草GE应用
随着基因编辑技术的普遍应用,CRISPR/Cas9系统已经成为烟草基因组编辑的基础工具。Xu等[70]利用CRISPR/Cas9介导NtPOD63 L(一种过氧化物酶)基因的敲除,发现NtPOD63 L敲除个体具有较高的耐旱性。Matsuo[71]以瞬时绿色荧光蛋白(GFP)基因表达监测基因编辑,利用CRISPR/Cas9成功敲除了本氏烟草的NbDCL2和NbDCL4基因,获得了双敲除烟草突变体,发现双敲除烟草突变体有可能作为重组蛋白生产的平台植物。Zhang等[72]利用CRISPR/Cas9敲除了烟草BBLs(烟碱合成相关基因),发现BBLs敲除突变体中,烟碱和大多数生物碱含量都明显减少,肌苷及其衍生物的含量急剧增加。随着对基因编辑效率要求的提升,CRISPR/Cas9被逐渐优化,Zhang等[73]使用OsU3-tRNA启动子组合代替AtU6启动子(OsU3-tRNA、AtU6均能启动sgRNA转录),并将AtUb10-Ros1表达盒融合到T-DNA上进行监测,对现有的烟草CRISPR/Cas9系统(pORE-Cas9)进行了优化,获得了新系统pOREU3TR,并在对烟草NtLHT1基因的定点编辑中证明了pOREU3TR的高效率。CRISPR/Cas9能够针对烟草基因组的特定区域进行精确地编辑,进而对某些特定的基因进行敲除、敲入或修饰,从而实现对烟草精准高效遗传改良。
5. 总结与展望
MAS已经通过QTL定位和GWAS设计了大量与烟草重要性状密切相关的分子标记,GS利用GEBV将对后代个体的选择简单化、数据化,GE能通过对基因的精确修饰快速实现对性状的定向改变,各种基于基因组学的育种技术的不断发展都将推动GAB在烟草育种上的发展和应用。建议从以下4个方面深化研究:
(1)充分利用烟草基因组信息。测序技术的进步和生物信息学的发展加快了烟草基因组的解析,但由于其多倍体的性质,基因组结构高度复杂,对烟草基因组的解析仍然面临许多技术难题。需进一步深入探究烟草基因组结构和功能,不断发展和完善利用烟草基因组的技术和方法,以便更好地支持烟草育种工作的开展。另外,相较单一的参考基因组,泛基因组涵盖更多的遗传多样性。Zhou等[74]和Tang等[75]首次解析了番茄和二倍体马铃薯的泛基因组,研究了茄科茄属的物种进化,为解析生物复杂性状的遗传机制提供了新思路。泛基因组在烟草上尚未开展研究,可能为烟草品种改良提供新思路。
(2)加快烟草分子标记的开发。对于植物分子标记的开发主要存在以下问题:①分子标记的筛选鉴定成本较高。②标记与性状之间的关联不明显或不稳定,导致选择的准确性降低。③MAS的累积效应受环境因素和遗传背景的影响较大,很难筛选到稳定的分子标记。建议从以下几个方面研究解决:①加快改进和完善自动化检测体系,建立DNA提取、PCR检测等移动工作站,实现科研自动化,减少消耗;②优化标记与性状之间的关联模型;③根据育种需求选择合适的群体进行分子标记的开发;④对于微效多基因的数量性状,应使用尽可能多的分子标记以提高选择的准确性。
(3)推进基因芯片在烟草育种上的应用。目前已经在烟草基因组上鉴定了大量的SNP,利用基因芯片技术可快速检测目标性状相关的SNP,加快优良个体的选择,但受到技术和成本限制,基因芯片在烟草育种实践中的应用相对较少。应加速完善基因芯片相关技术,降低使用成本,以更好地支撑和促进烟草育种工作的进步。
(4)加强烟草高通量表型监测平台的建设。目前作物表型获取主要是依靠人力,耗时耗力且数量获取有限,无法提供准确预测复杂数量性状所需的大规模、高质量的表型数据,而高通量表型监测平台将有望解决这一难题,应加强烟草高通量表型平台的建设。烟草高通量表型与高通量遗传信息的结合,将为烟草的定向改良带来更精确的育种策略。
-
图 1 烟草基因组辅助育种流程图[4]
Figure 1. Flowchart for genome-assisted breeding in tobacco
-
[1] JIAO Q, DENG J H, ZHAO X Y, et al . Physiological and biochemical regulation of tobacco by oxathiapiprolin under Phytophthora nicotianae infection[J]. Physiologia Plantarum,2023 ,175 (2 ):1 -16 .[2] MARQUES T L, PADUA J M V, BERGER I J, et al . Strategies for the recurrent selection program in tobacco breeding for green leaf yield[J]. Crop Science,2022 ,62 (6 ):2212 -2221 . doi: 10.1002/csc2.20813[3] LEE P N, FRY J S . Relating onset of health conditions to changes in tobacco/nicotine use-analyses based on the US PATH study[J]. Contributions to Tobacco & Nicotine Research,2023 ,32 (1 ):1 -10 .[4] LENG P, THOMAS L, XU M . Genomics-assisted breeding-A revolutionary strategy for crop improvement[J]. Journal of Integrative Agriculture,2017 ,16 (12 ):2674 -2685 . doi: 10.1016/S2095-3119(17)61813-6[5] JANGRA S, CHAUDHARY V, YADAV R C, et al . High-throughput phenotyping: a platform to accelerate crop improvement[J]. Phenomics,2021 ,1 (2 ):31 -53 . doi: 10.1007/s43657-020-00007-6[6] ZAN Y J, CHEN S, REN M, et al. The allotetraploid Nicotiana tabacum genome and GenBank genomics highlight the genomic features, genetic diversity and regulation of morphological, metabolic and disease-resistance traits[J]. BioRxiv, 2023.
[7] EATHINGTON S R, CROSBIE T M, EDWARDS M D, et al . Molecular markers in a commercial breeding program[J]. Crop Science,2007 ,47 :154 -163 .[8] COBB J N, BISWAS P S, PLATTEN J D . Back to the future: revisiting MAS as a tool for modern plant breeding[J]. Theor Appl Genet.,2019 ,132 (3 ):647 -667 . doi: 10.1007/s00122-018-3266-4[9] SINGH G, SINGH N, ELLUR R K, et al . Genetic enhancement for biotic stress resistance in basmati rice through marker-assisted backcross breeding[J]. International Journal of Molecular Sciences,2023 ,24 (22 ):16081 . doi: 10.3390/ijms242216081[10] JAMALODDIN M, RANI, C V D, SWATHI G, et al . Marker assisted gene pyramiding (MAGP) for bacterial blight and blast resistance into mega rice variety ‘Tellahamsa’[J]. PLoS One,2020 ,15 (6 ):e0234088 . doi: 10.1371/journal.pone.0234088[11] THOMSON J M . High-throughput SNP genotyping to accelerate crop improvement[J]. Plant Breeding and Biotechnology,2014 ,2 (3 ):195 -212 . doi: 10.9787/PBB.2014.2.3.195[12] TONG Z J, XIU Z H, MING Y, et al. Quantitative trait locus mapping and genomic selection of tobacco (Nicotiana tabacum L.) based on high-density genetic map[J]. Plant Biotechnology Reports, 2021, 15(6): 845-854.
[13] 曾婉俐, 梁岗, 高茜, 等. 烟草香气相关基因CRISPR/Cas9编辑突变体库的构建[J/OL]. 广西植物. [2023-07-09]. http://kns.cnki.net/kcms/detail/45.1134.q.20231206.1136.002.html. ZENG W L, LIANG G, GAO X, et al. Construction of a CRISPR/Cas9 edited mutant library of tobacco aroma-related genes[J]. Guangxi Plant. [2023-07-09]. http://kns.cnki.net/kcms/detail/45.1134.q.20231206.1136.002.html.
[14] JIARUI J R, HAITAO H T, QIAN G, et al . Effects of editing DFR genes on flowers, leaves, and roots of tobacco[J]. BMC Plant Biology,2023 ,23 (1 ):349 -349 . doi: 10.1186/s12870-023-04307-7[15] LI, G M, MA, Y X, WANG, X P, et al . CRISPR/Cas9 gene editing of NtAITRs, a family of transcription repressor genes, leads to enhanced drought tolerance in tobacco[J]. International Journal of Molecular Sciences,2022 ,23 (23 ):15268 -15268 . doi: 10.3390/ijms232315268[16] CLARKSON J J, LIM K Y, KOVARIK A, et al . Long-term genome diploidization in allopolyploid nicotiana section repandae (Solanaceae)[J]. New Phytol.,2005 ,168 (1 ):241 -252 . doi: 10.1111/j.1469-8137.2005.01480.x[17] 国家烟草专卖局. 绒毛状烟草和林烟草全基因组序列图谱完成[EB/OL]. [2023-07-09]. http://www.tobacco.gov.cn/html/30/3004/3893491_n.html. State Tobacco Monopoly Administration. Whole genome sequence mapping of downy and forest tobacco completed [EB/OL]. [2023-07-09]. http://www.tobacco.gov.cn/html/30/3004/3893491_n.html.
[18] SIERRO N, BATTEY J N D, OUADI S, et al . Reference genomes and transcriptomes of Nicotiana sylvestris and Nicotiana Tomentosiformis[J]. Genome Biology,2013 ,14 (6 ):1 -17 .[19] SIERRO N, BATTEY J N D, OUADI S, et al . The tobacco genome sequence and its comparison with those of tomato and potato[J]. Nature Communications,2014 ,5 (1 ):1 -9 .[20] KUROTANI K I, HIRAKAWA H, SHIRASAWA K, et al . Genome sequence and analysis of Nicotiana benthamiana, the model plant for interactions between organisms[J]. Plant and Cell Physiology,2023 ,64 (2 ):248 -257 . doi: 10.1093/pcp/pcac168[21] XU S Q, BROCKMöLLER T, NAVARRO-QUEZADA A, et al . Wild tobacco genomes reveal the evolution of nicotine biosynthesis[J]. Proceedings of the National Academy of Sciences,2017 ,114 (23 ):6133 -6138 . doi: 10.1073/pnas.1700073114[22] SIERRO N, BATTEY J N D, BOVET L, et al . The impact of genome evolution on the allotetraploid Nicotiana rustica-an intriguing story of enhanced alkaloid production[J]. BMC Genomics,2018 ,19 (1 ):1 -18 . doi: 10.1186/s12864-017-4368-0[23] WANG L, XIE X D, XU Y L, et al . Comprehensive analysis of the carboxylesterase gene reveals that NtCXE22 regulates axillary bud growth through strigolactone metabolism in tobacco[J]. Frontiers in Plant Science,2022 ,13 :1019539 .[24] ZHANG X Q, WEI X, WANG M P, et al . Overexpression of NtabDOG1L promotes plant growth and enhances drought tolerance in Nicotiana tabacum[J]. Plant Science,2019 ,287 :110186 . doi: 10.1016/j.plantsci.2019.110186[25] MAO J J, YUAN G, HAN K, et al . Genome-wide identification of CBL family genes in Nicotiana tabacum and the functional analysis of NtCBL4A-1 under salt stress[J]. Environmental and Experimental Botany,2023 ,209 :105311 . doi: 10.1016/j.envexpbot.2023.105311[26] ZHANG L Y, XU Z Q, JIANG Z M, et al. Cloning and functional analysis of the root-knot nematode resistance gene NtRk1 in tobacco[J]. Physiol Plant, 2023: e13894.
[27] SUI X, HE X, SONG Z, et al . The gene NtMYC2a acts as a ‘master switch’ in the regulation of JA induced nicotine accumulation in tobacco[J]. Plant Biology,2021 ,23 (2 ):317 -326 . doi: 10.1111/plb.13223[28] BURNER N, KERNODLE S P, STEEDE T, et al . Editing of A622 genes results in ultra-low nicotine whole tobacco plants at the expense of dramatically reduced growth and development[J]. Molecular Breeding,2022 ,42 (4 ):20 -30 . doi: 10.1007/s11032-022-01293-w[29] COLLARD B C Y, MACKILL D J . Marker-assisted selection: an approach for precision plant breeding in the twenty-first century[J]. Philosophical Transactions of the Royal Society B:Biological Sciences,2007 ,363 (1491 ):557 -572 .[30] JULIANA P, POLAND J, HUERTA-ESPINO J, et al . Improving grain yield, stress resilience and quality of bread wheat using large-scale genomics[J]. Nature Genetics,2019 ,51 (10 ):1530 -1539 . doi: 10.1038/s41588-019-0496-6[31] LIN T Y, KAO Y Y, LIN S, et al . A genetic linkage map of Nicotiana plumbaginifolia/Nicotiana longiflora based on RFLP and RAPD markers[J]. Theor Appl Genet,2001 ,103 :905 -911 . doi: 10.1007/s001220100618[32] NISHI T, TAJIMA T, NOGUCHI S, et al . Indentification of DNA markers of tobacco linked to bacterial wilt resistance[J]. Theor Appl Genet,2003 ,106 :700 -705 .[33] XIAO B G, TAN Y T, LONG N, et al . SNP-based genetic linkage map of tobacco (Nicotiana tabacum L. ) using next-generation RAD sequencing[J]. J Biol Res (Thessalon),2015 ,22 :1 -11 . doi: 10.1186/s40709-015-0025-4[34] TONG Z J, ZHOU J H, XIU Z H, et al. Construction of a high-density genetic map with whole genome sequencing in Nicotiana tabacum L[L]. Genomics, 2020, 112(2): 2028-2033.
[35] HILL W G, MACKAY T F . D. S. Falconer and introduction to quantitative genetics[J]. Genetics,2004 ,167 :1529 -1536 . doi: 10.1093/genetics/167.4.1529[36] WEI J L, XU S Z . A random-model approach to QTL mapping in multiparent advanced generation intercross (MAGIC) populations[J]. Genetics,2016 ,202 (2 ):471 -486 . doi: 10.1534/genetics.115.179945[37] LI G, ZHOU Y H, LI H F, et al . A multi-locus linear mixed model methodology for detecting small-effect QTLs for quantitative traits in MAGIC, NAM, and ROAM populations[J]. Computational and Structural Biotechnology Journal,2023 ,21 :2241 -2252 . doi: 10.1016/j.csbj.2023.03.022[38] ZHANG Z, ERSOZ E, LAI C Q, et al . Mixed linear model approach adapted for genome-wide association studies[J]. Nature Genet,2010 ,42 :355 -360 . doi: 10.1038/ng.546[39] 赵会纳, 雷波, 程立锐, 等 . 烟草叶宽性状主效QTL定位及育种评价[J]. 中国烟草科学,2023 ,44 (1 ):1 -7 .ZHAO H N, LEI B, CHENG L R, et al . Localization and breeding evaluation of QTL for leaf width trait in tobacco[J]. Chinese Tobacco Science,2023 ,44 (1 ):1 -7 .[40] 刘颖超, 方敦煌, 徐海明, 等 . 烟草生物碱性状的QTL定位[J]. 作物学报,2024 ,50 (1 ):42 -54 .LIU Y C, FANG D H, XU H M, et al . QTL localization of tobacco alkaloid traits[J]. Acta Agronomica Sinica,2024 ,50 (1 ):42 -54 .[41] AGACKA-MOłDOCH M, ARIF M A R, LOHWASSER U, et al . QTL analysis of seed germination traits in tobacco (Nicotiana tabacum L. )[J]. J. Appl. Genet,2021 ,62 :441 -444 . doi: 10.1007/s13353-021-00623-6[42] 姜自鹏, 赵会纳, 苑广迪, 等 . 烟草株高和叶数性状QTL定位及候选基因预测[J]. 中国烟草科学,2022 ,43 (2 ):1 -6 .JIANG Z P, ZHAO H N, YUAN G D, et al . Localization of QTL and prediction of candidate genes for tobacco plant height and leaf number[J]. Chinese Tobacco Science,2022 ,43 (2 ):1 -6 .[43] 蒋勋, 宋健, 刘国祥, 等 . 雪茄烟遗传图谱构建及叶面重要性状的QTL定位[J]. 分子植物育种,2022 ,20 (18 ):6076 -6086 .JIANG X, SONG J, LIU G X, et al . Construction of a genetic map of cigar tobacco and localization of QTL for important foliar traits[J]. Molecular Plant Breeding,2022 ,20 (18 ):6076 -6086 .[44] TONG Z J, XU M L, ZHANG Q X, et al . Construction of a high-density genetic map and dissection of genetic architecture of six agronomic traits in tobacco (Nicotiana tabacum L. )[J]. Frontiers in Plant Science,2023 ,14 :1126529 . doi: 10.3389/fpls.2023.1126529[45] YUAN G D, SUN K F, YU W L, et al . Development of a MAGIC population and high-resolution quantitative trait mapping for nicotine content in tobacco[J]. Frontiers in Plant Science,2023 ,13 :1086950 . doi: 10.3389/fpls.2022.1086950[46] 王思齐, 李海洋, 李荣华, 等 . 烟草青枯病抗病的动态QTL分析[J]. 中国烟草科学,2020 ,41 (3 ):1 -8 .WANG S Q, LI H Y, LI R H, et al . Dynamic QTL analysis of resistance to tobacco greening blight[J]. Chinese Tobacco Science,2020 ,41 (3 ):1 -8 .[47] MA J M, HEIM C, HUMPHRY M, et al . Genetic analysis of Phn7.1, a major QTL conferring partial resistance to Phytophthora nicotianae in Nicotiana tabacum[J]. Molecular Breeding,2019 ,39 (1 ):11 -26 . doi: 10.1007/s11032-018-0923-x[48] WANG M Y, XU S Z . Statistical power in genome-wide association studies and quantitative trait locus mapping[J]. Heredity,2019 ,123 (3 ):287 -306 .[49] LIU X, HUANG M, FAN B, et al . Iterative usage of fixed and random effect models for powerful and efficient genome-wide association studies[J]. PLoS genetics,2016 ,12 (2 ):e1005767 . doi: 10.1371/journal.pgen.1005767[50] KALER A S, GILLMAN J D, BEISSINGER T, et al . Comparing different statistical models and multiple testing corrections for association mapping in soybean and maize[J]. Frontiers in Plant Science,2020 ,10 :1794 . doi: 10.3389/fpls.2019.01794[51] 孙滢, 姜自鹏, 刘洪泰, 等 . 烟草开花期全基因组关联分析[J]. 中国烟草科学,2020 ,41 (6 ):1 -6 .SUN Y, JIANG Z P, LIU H T, et al . Genome-wide association analysis of tobacco flowering time[J]. China Tobacco Science,2020 ,41 (6 ):1 -6 .[52] IKRAM M, XIAO J W, LI R H, et al . Identification of superior haplotypes and candidate genes for yield-related traits in tobacco (Nicotiana tabacum L. ) using association mapping[J]. Industrial Crops and Products,2022 ,189 :115886 . doi: 10.1016/j.indcrop.2022.115886[53] IKRAM M, LAI R Q, XIA Y S, et al . Genetic dissection of tobacco (Nicotiana tabacum L. ) plant height using single-locus and multi-locus genome-wide association studies[J]. Agronomy,2022 ,12 (5 ):1047 . doi: 10.3390/agronomy12051047[54] TONG Z J, FANG D H, CHEN X J, et al . Genome-wide association study of leaf chemistry traits in tobacco[J]. Breeding Science,2020 ,70 (3 ):253 -264 . doi: 10.1270/jsbbs.19067[55] XU X, WANG Z, XU S X, et al . Identifying loci controlling total starch content of leaf in Nicotiana tabacum through genome-wide association Study[J]. Functional & Integrative Genomics,2022 ,22 (4 ):537 -552 .[56] MANSFIELD J, GENIN S, MAGORI S, et al . Top 10 plant pathogenic bacteria in molecular plant pathology[J]. Molecular plant pathology,2012 ,13 (6 ):614 -629 . doi: 10.1111/j.1364-3703.2012.00804.x[57] LAI R Q, IKRAM M, LI R H, et al . Identification of novel quantitative trait nucleotides and candidate genes for bacterial wilt resistance in tobacco (Nicotiana tabacum L. ) using genotyping-by-sequencing and multi-locus genome-wide association studies[J]. Frontiers in Plant Science,2021 ,12 :744175 . doi: 10.3389/fpls.2021.744175[58] 何斌彬, 耿锐梅, 杨爱国, 等 . 烟草青枯病抗性的全基因组关联分析[J]. 中国烟草科学,2020 ,41 (5 ):1 -7 .HE B B, GENG R M, YANG A G, et al . Genome-wide association study of resistance to tobacco bacterial wilt[J]. Chinese Tobacco Science,2020 ,41 (5 ):1 -7 .[59] GUO T, YANG J, LI D X, et al . Integrating GWAS, QTL, mapping and RNA-seq to identify candidate genes for seed vigor in rice (Oryza sativa L. )[J]. Molecular Breeding,2019 ,39 (6 ):87 -103 . doi: 10.1007/s11032-019-0993-4[60] WANG X, XU Y, HU Z L, et al. Genomic selection methods for crop improvement: current status and prospects[J]. Elsevier BV, 2018: 330-340.
[61] WANG B B, LIN Z C, LI X, et al . Genome-wide selection and genetic improvement during modern maize breeding[J]. Nature Genetics,2020 ,52 (6 ):565 -571 . doi: 10.1038/s41588-020-0616-3[62] LORENZ A J, SMITH K P. Adding genetically distant individuals to training populations reduces genomic prediction accuracy in barley[J]. Wiley, 2015: 2657-2667.
[63] NIELSEN N H, JAHOOR A, JENSEN J D, et al. Genomic prediction of seed quality traits using advanced barley breeding lines[J]. Public Library of Science, 2016: e0164494.
[64] MEUWISSEN T H E . Accuracy of breeding values of “unrelated” individuals predicted by dense SNP genotyping[J]. Genetics Selection Evolution,2009 ,41 (1 ):35 -44 . doi: 10.1186/1297-9686-41-35[65] MEUWISSEN T H E, HAYES B J, GODDARD M E . Prediction of total genetic value using genome-wide dense marker maps[J]. Genetics,2001 ,157 :1819 -1829 . doi: 10.1093/genetics/157.4.1819[66] TONG Z J, XIU Z H, MING Y, et al . Quantitative trait locus mapping and genomic selection of tobacco (Nicotiana tabacum L. ) based on high-density genetic map[J]. Plant Biotechnology Reports,2021 ,15 (6 ):845 -854 . doi: 10.1007/s11816-021-00713-1[67] CARVALHO B L, LEWIS R, BRUZI A T, et al . Adding genome‐wide genotypic information to a tobacco (Nicotiana tabacum L. ) breeding programme[J]. Plant Breeding,2022 ,141 (1 ):133 -141 . doi: 10.1111/pbr.12979[68] JIANG F J, DOUDNA J A . CRISPR–Cas9 structures and mechanisms[J]. Annual Review of Biophysics,2017 ,46 :505 -529 . doi: 10.1146/annurev-biophys-062215-010822[69] CONG L, RAN F A, COX D, et al . Multiplex genome engineering using CRISPR/Cas systems[J]. Science,2013 ,339 (6121 ):819 -823 . doi: 10.1126/science.1231143[70] XU L, GAO Q, FENG J Y, et al . Physiological and phosphoproteomic analyses revealed that the NtPOD63 L knockout mutant enhances drought tolerance in tobacco[J]. Industrial Crops and Products,2023 ,193 :116218 . doi: 10.1016/j.indcrop.2022.116218[71] MATSUO K . CRISPR/Cas9-mediated knockout of the DCL2 and DCL4 genes in Nicotiana benthamiana and its productivity of recombinant proteins[J]. Plant Cell Reports,2021 ,41 (2 ):307 -317 .[72] ZHANG J D, ZHOU Q, ZHANG D H, et al . The agronomic traits, alkaloids analysis, FT-IR and 2DCOS-IR apectroscopy identification of the low-nicotine-content nontransgenic tobacco edited by CRISPR–Cas9[J]. Molecules,2022 ,27 (12 ):3817 . doi: 10.3390/molecules27123817[73] ZHANG J D, XING J X, MI Q L, et al . Highly efficient transgene-free genome editing in tobacco using an optimized CRISPR/Cas9 system, POREU3TR[J]. Plant Science,2023 ,326 :111523 . doi: 10.1016/j.plantsci.2022.111523[74] ZHOU Y, ZHANG Z Y, BAO Z G, et al . Graph pangenome captures missing heritability and empowers tomato breeding[J]. Nature,2022 ,606 (7914 ):527 -534 . doi: 10.1038/s41586-022-04808-9[75] TANG D, JIA Y X, ZHANG J Z, et al . Genome evolution and diversity of wild and cultivated potatoes[J]. Nature,2022 ,606 (7914 ):535 -541 . doi: 10.1038/s41586-022-04822-x -
期刊类型引用(2)
1. 周昆燕,徐兴阳,刘霞,杨艳丽,端永明,胡先奇. 烟草疫霉侵害对嫁接烟株叶片相关防御酶活性和MDA含量的影响. 江西农业学报. 2025(01): 65-72 .  百度学术
百度学术
2. 吴孟婷,籍燕,周东新,刘国祥,燕思凡,李媛,宁扬,林天然,李洋洋,张兴伟,程崖芝. 烟草SAUR基因家族鉴定及表达分析. 中国烟草科学. 2024(06): 17-26+38 .  本站查看
本站查看
其他类型引用(0)



 下载:
下载: